|
イカ塩辛の細菌叢解析法
北里大学海洋生命科学部
特別研究補助員 水澤 奈々美 客員教授 渡部 終五 1. イカ塩辛における細菌叢管理の重要性我が国における発酵食品には、味噌、醤油、酒、味醂をはじめとする様々なものが挙げられるが、塩辛は、代表的な水産発酵食品の一つである。日本各地で様々な塩辛が作られており、カツオの塩辛(酒盗、ワタガラス)、鮎の生殖腺や内臓、身などを用いた塩辛(うるか)、ナマコの内臓の塩辛(このわた)、鮭の腎臓の塩辛(めふん)など枚挙にいとまがない1)。その中でも、最も有名なものは、イカの塩辛であろう(写真)。
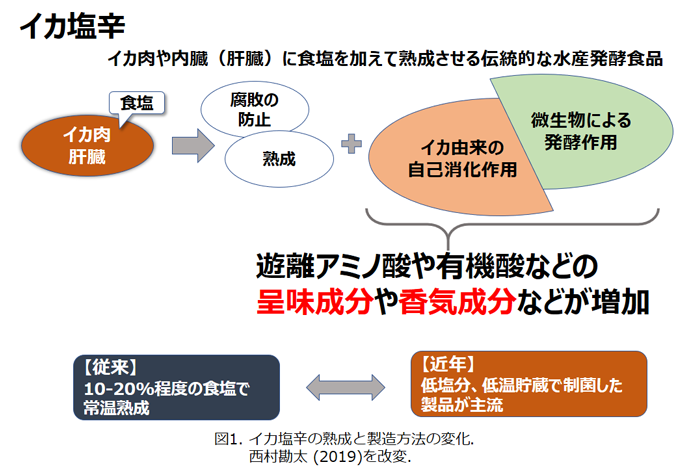
伝統的なイカ塩辛の製造方法には、赤作り、白作り、黒作りなど様々なものがあるが、基本的には、イカ肉に肝臓と塩を加え常温で熟成させたものである。さらに、これらに糀を添加したものもある。このような伝統的イカ塩辛の熟成には、微生物による発酵と、原料であるイカの筋肉や内臓由来の消化酵素による自己消化作用が重要な役割を果たす(図1)。
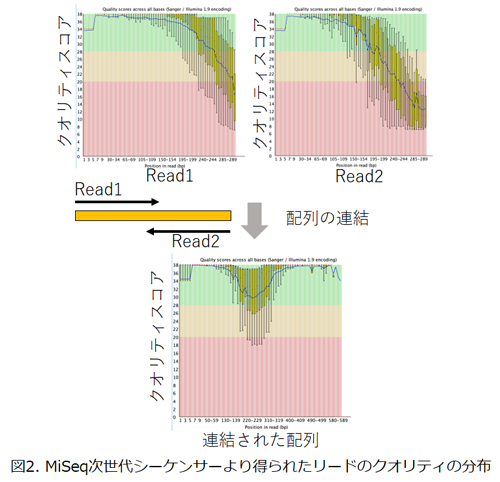
微生物による発酵作用と内在性の酵素による自己消化作用がイカ塩辛の発酵にどのように寄与しているかは長らく議論の的であったが、近年では、自己消化作用が旨味に関与する遊離アミノ酸の生成に2)、微生物の発酵作用により生成した有機酸などが独特の風味の醸成に関わっていることが明らかとなってきた3)。このような伝統的塩辛では、熟成が数日間と急速に進むため発酵の制御が難しく、その品質管理は職人の技に依存するとされる。 一方、近年の消費者の健康志向の高まりから、低塩分の塩辛も多く流通している。低塩分の塩辛では、塩分が低いため、熟成中の食中毒菌の増殖を抑えることができない。このため、常温による熟成が行えず、調味や防腐、離水防止などの目的で多種類の添加物を添加し作製される4)。また、低塩分塩辛では作製から流通まで徹底した低温管理が求められるが、食中毒事例も報告されている。このように、イカ塩辛では細菌叢の制御は品質管理や安全管理において非常に重要であることから、著者らはイカ塩辛の細菌叢解析を行ってきた。 従来の微生物研究では、試料中に含まれる微生物を培養することによって、そこに含まれる種を同定する手法がとられてきた。近年、次世代シーケンサー(NGS)の登場によって、数百万から数十億もの膨大な遺伝子の配列解析を同時に行うことが可能となった。これにより、従来の培地培養では検出できなかった難培養性細菌を含めた様々な細菌の存在を、DNAの解析で簡便に検出することができるようになった。食品や医療など、様々な分野において現在広く行われている微生物叢解析は、NGSによるものが主流であると言っても過言ではないだろう。このNGSを用いた細菌叢解析(メタゲノム解析)を行うにあたり、高品質なDNAを試料から調製することが求められるほか、DNA解析に用いる遺伝子領域の検討などが必要となる。本記事では、著者らがイカ塩辛のNGSを用いた微生物叢解析について検討を行った結果を紹介したい。 2. イカ塩辛のDNA抽出法の検討NGSに供するDNAは高品質であることが求められる。そこで、近年多く流通している低塩分塩辛と、DNA抽出が難しいと考えられる糀を含んだ高塩分の伝統的イカ塩辛(高塩分塩辛)を試料として、最適なDNA抽出法を検討した。市販の糞便からのDNA抽出キット、土壌からのDNA抽出キット、および臭化ヘキサデシルトリメチルアンモニウム(CTAB) 法を用いて検討を行った。CTAB法は植物からのDNA抽出によく用いられる方法であり、多糖の除去に優れるとされる5)。高塩分塩辛では糀由来の多量の多糖が含まれることが予想されたため、この方法を候補として選択した。 低塩分塩辛および高塩分塩辛を試料として、糞便および土壌からのDNA抽出キットを用いて抽出したDNA溶液では、核酸に特徴的な260 nmのピークは認められなかった。一方、CTAB法で抽出したDNA溶液では260 nmに明確なピークが認められた。従って、CTAB法により抽出したDNA溶液の方が、市販キットで抽出したDNA溶液より純度が優れていると考えられた6)。そこで、イカ塩辛のDNA抽出には、CTAB法を用いることとした。 3. 細菌叢解析に用いる16S rRNA遺伝子領域の検討細菌叢解析には16S rRNA遺伝子を用いた。この遺伝子は、細菌叢解析によく用いられる遺伝子の一つであり、種間で塩基配列がよく保存されている保存領域と、種ごとに塩基配列が大きく異なるV1~V9の9個の可変領域からなる。可変領域は、細菌によって検出のされやすさが異なる場合があるため、複数領域を比較し、解析にどの領域を用いるかを決定する必要がある。そこで著者らはイカ塩辛を対象に、解析に用いる可変領域の検討を行った。解析に用いられる可変領域は研究対象試料により様々であるが、著者らは16S rRNA遺伝子のV1-V2、V1-V3、V3、V3-V4およびV4-V5領域を検討した。 MiSeq次世代シーケンサーは、5’および3’ 末端の両端より300 bpを解析するペアエンド法により約600 bpの解析が可能である。次世代シーケンサーから得られる各塩基には、確からしさを表すクオリティ(Q)スコアが付けられている。それぞれの塩基についたQスコアは、たとえばQ20なら99%、Q30なら99.9%の確からしさとして表される。MiSeqでは、解析塩基長の増加に伴い末端付近の部分のクオリティが低下することが知られている。このようなクオリティの低くなる部分でも、5’および3’両端から解析された配列を連結させることにより、お互いに補完してクオリティを一定以上に保つことができるようになっている(図2)。しかしながら、実際の解析を行う際には、より信頼性の高い結果を得るために、さらにこのクオリティの調整が必要となる7)。
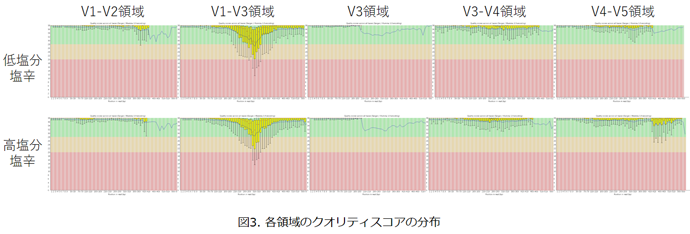
低塩分塩辛と高塩分塩辛について、16S rRNA遺伝子のそれぞれの可変領域についてPCR増幅したDNAをMiSeqに供した。低塩分塩辛、高塩分塩辛ともに、V1-V2領域、V3領域およびV3-V4領域ではクオリティスコアの低い塩基配列部分は認められなかった(図3)。
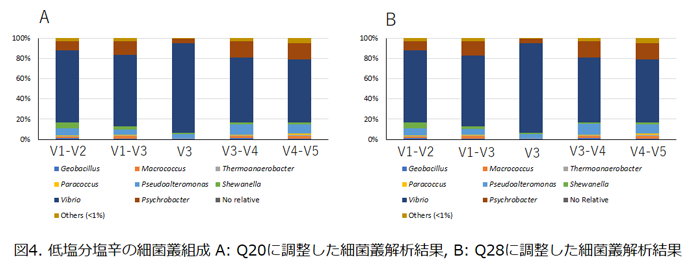
一方、低塩分塩辛、高塩分塩辛ともにV1-V3領域と、高塩分塩辛ではV4-V5領域においてもクオリティスコアが顕著に低い部分が認められた。 V1-V3領域の塩基配列長は約520 bpと他の領域に比べて長く、元々クオリティが顕著に低い部分を連結しても改善されなかったと考えられる。 これらの各可変領域につき、クオリティスコアの調整による細菌叢解析結果への影響を検討した。クオリティスコアを20以上に調整し細菌叢解析を行った場合では、低塩分塩辛では全ての可変領域でVibrio属が優占菌であったが、とくにV3領域でVibrio属の組成比が高かった。クオリティスコアを28以上に調整した場合でも、解析結果はほとんど変わらなかった(図4)。
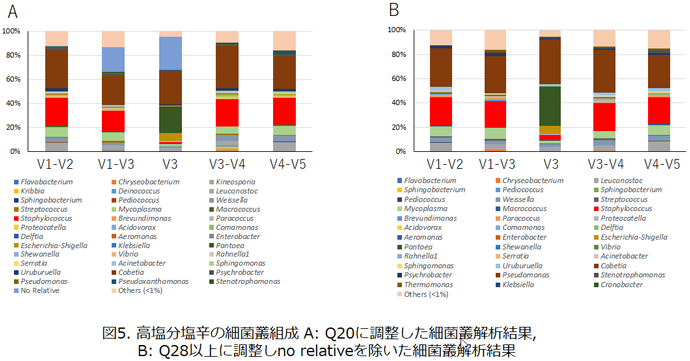
一方、高塩分塩辛では、イカ塩辛の作製時に材料として添加した米糀由来の配列がいずれの領域でも検出された。そこで、これらの配列を除去したのち、クオリティスコアを20以上に調整して細菌叢解析を行った場合、V1-V3およびV3領域で公表されているデーターベースにヒットしないno relativeの塩基配列(リード)が20-30%程度認められた(図5A)。一方、クオリティスコアを28以上に調整した場合では、V3領域ではno relativeと判定されるリードが認められなくなった。従って、V3領域では、クオリティの低い配列がno relativeと判定されていたと考えられる。一方、V1-V3領域においてもクオリティスコアを28以上に調整したところno relativeのリードは大きく減少したことから、クオリティスコア20以上で解析して得られたno relativeは低クオリティのリードに由来すると考えられた。V1-V3領域でこのリードを除去したところ、高塩分塩辛ではV1-V2領域、V3-V4領域およびV4-V5領域の解析結果と同様にCobetia属とStaphylococcus属が優占菌となった(図5B)。
一方、V3領域の解析ではPantoea属とCobetia属が優占菌となり、他の可変領域の解析とは異なった。なお、著者らのイカ塩辛の細菌叢解析結果の一部は既に発表済みである6, 8-12)。 最後に以上の結果から、MiSeq次世代シーケンサーを用いたイカ塩辛の細菌叢解析に有効な16S rRNA遺伝子の解析領域は、V1-V2領域、V3-V4領域またはV4-V5領域であると考えられた。V1-V3領域についてもno relativeのリードの除去により、V1-V2領域、V3-V4領域およびV4-V5領域と類似した解析結果を示し、適切なクオリティスコアの調整を施せば利用可能と考えられた。このように、解析に用いる塩基配列長およびクオリティスコアの調整は解析結果に大きく影響することが示された。したがって、他の研究対象試料の細菌叢解析でも、解析領域、解析塩基長、リードのクオリティには注意が必要であると考えられる。 引用文献
略歴
水澤奈々美 北里大学・海洋生命科学部・特別研究補助員
東京大学大学院博士課程修了 博士(農学) 水産総合研究センター中央水産研究所 研究等支援職員 北里大学海洋生命科学部 特別研究補助員 北里大学海洋生命科学部 特任助教
渡部終五 北里大学・海洋生命科学部・客員教授
東京大学大学院農学系研究科博士課程修了(農学博士) 財団法人海洋生物環境研究所研究員 東京大学農学部助手 東京大学農学部助教授 東京大学大学院農学生命科学研究科教授 北里大学海洋生命科学部教授 サナテックメールマガジンへのご意見・ご感想を〈e-magazine@mac.or.jp〉までお寄せください。 |
| Copyright (C) Food Analysis Technology Center SUNATEC. All Rights Reserved. |